 >
>
 >
>
La Medicina de Precisión propone incorporar la consideración de la variabilidad individual, especialmente la relacionada con los genes de cada persona en los métodos de prevención, diagnóstico y tratamiento, buscando maximizar su eficacia. De esta forma se logra que, para grupos determinados de pacientes (con la consideración de ciertos datos sobre sus exposiciones ambientales y elecciones de estilo de vida), se llegue a diagnósticos más precisos o se puedan definir mejores tratamientos y estrategias de prevención para una enfermedad particular.
Dos hitos fundamentales que permiten acercar la Medicina de Precisión a la práctica clínica fueron el Proyecto Genoma Humano (PGH) y el surgimiento de la secuenciación de segunda generación, secuenciación masiva o Next-Generation Sequencing (NGS). En el año 2003, con la finalización del PGH y la publicación de la secuencia esencial del genoma humano, comenzó el desarrollo y la traslación de distintos tipos de estudios genómicos, orientados a mejorar el conocimiento de los mecanismos moleculares de las enfermedades. Por otro lado, en el año 2007, la llegada de la tecnología de secuenciación masiva de ADN logró la disminución de costos de secuenciación (Figura 1), lo que aumentó de manera exponencial el conocimiento respecto de las causas genéticas relacionadas tanto con enfermedades extremadamente raras como con trastornos comunes pero heterogéneos, y favoreció un notable incremento de los estudios genéticos disponibles para los pacientes.
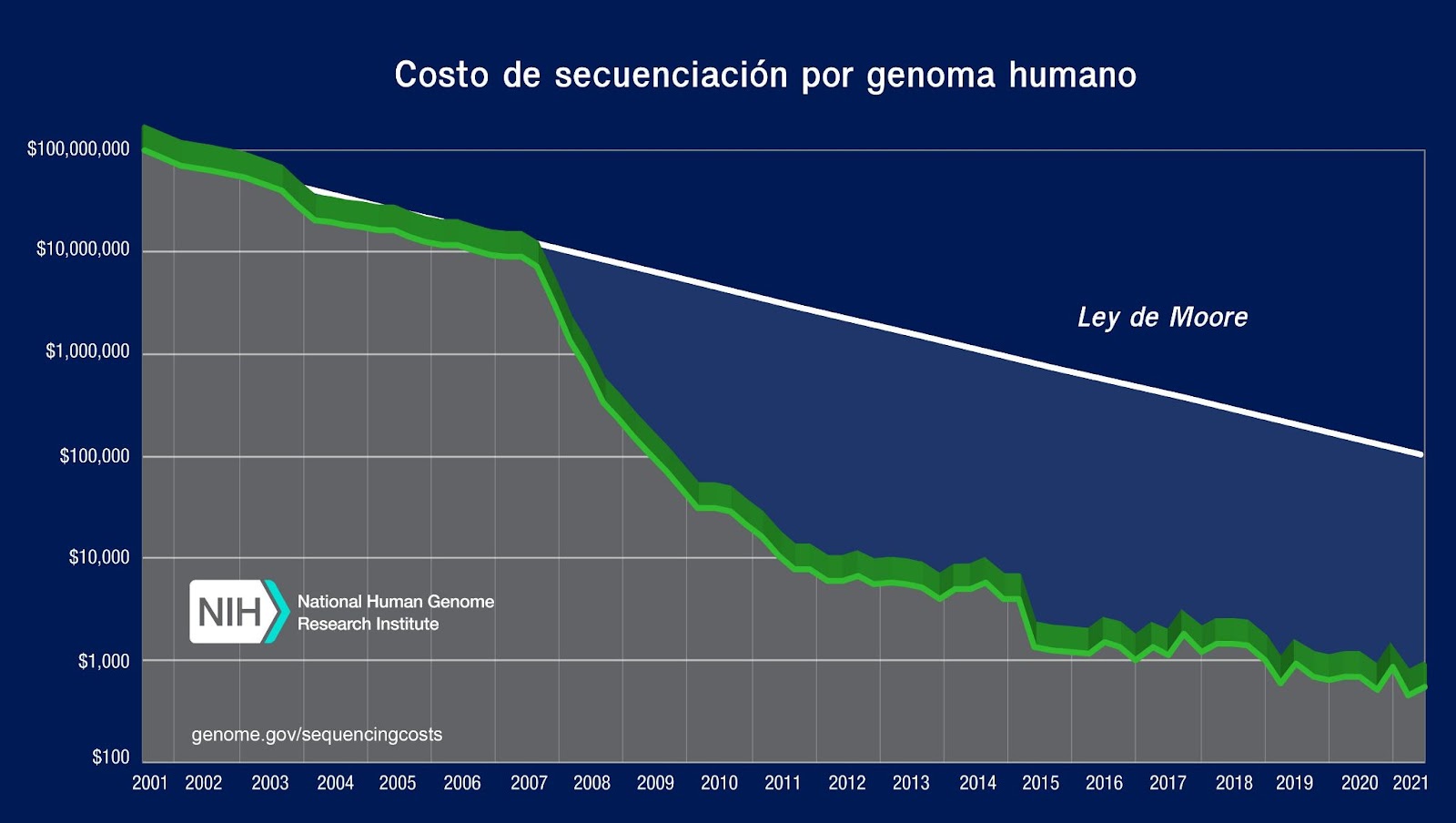
Figura 1: Costos de secuenciación del genoma humano. Fuente: Instituto Nacional de Salud de Estados Unidos (National Institutes of Health)
La aplicación de tecnologías de NGS trajo a su vez, nuevos desafíos informáticos para los laboratorios de biología molecular, ya que el volumen de datos que genera necesita de una gran infraestructura, de procesos ordenados, seguros y de recursos humanos bioinformáticos que sean capaces de definir cómo procesarlos y almacenarlos y de aportar las herramientas necesarias para colaborar con su análisis e interpretación.
La implementación en el hospital
Para hacer oportuno el uso de estos datos genómicos que se generan en un estudio genético y teniendo en cuenta la complejidad para interpretarlos, es necesario disponer de sistemas que integren diferentes fuentes de datos en la Historia Clínica Electrónica (HCE) y en los sistemas de Soporte a la Toma de Decisiones Clínicas (CDSS). De esta manera, se puede brindar el apoyo a los profesionales de la asistencia médica en la toma de decisiones en tiempo real, mejorando la capacidad para impactar positivamente en los resultados de los pacientes. Siguiendo esta premisa, el equipo de Bioinformática del Departamento de Informática en Salud del Hospital Italiano de Buenos Aires (HIBA), en colaboración con la empresa de Medicina de Precisión genomIT, ha trabajado en el diseño, desarrollo e implementación de un ecosistema informático que permite la limpieza y el almacenamiento de los datos genómicos de los pacientes del HIBA, el enriquecimiento de los mismos con bases de datos bioinformáticas y la integración con el resto de los sistemas de información en salud de la institución.
El proyecto comenzó a mediados del año 2019, con la idea principal de incorporar bases de datos genéticas que permitan enriquecer el contenido de la HCE, con información genómica y realizar sugerencias de riesgo genético y seguimiento a través del CDSS. El equipo interdisciplinario que trabaja en el proyecto integra médicos genetistas, bioinformáticos, informáticos clínicos y desarrolladores e ingenieros de software de ambas instituciones.
El ecosistema bioinformático fue implementado en el HIBA, institución que cuenta con un laboratorio de secuenciación de ADN y con un sistema de información de salud de desarrollo propio desde 1998. Este sistema está certificado por la Sociedad de Sistemas de Información y Gestión de la Salud (HIMSS, del inglés Healthcare Information and Management Systems Society), con el nivel más alto en el Modelo de Adopción de Historia Clínica Electrónica.
La arquitectura de la solución incluye bases de datos genómicas que contienen los datos de los pacientes y bases de datos bioinformáticas públicas integradas localmente. La información alojada en estas bases de datos puede ser accedida por diversos clientes -incluídos la HCE y CDSS- a través de una API basada en nomenclaturas y estándares de interoperabilidad en salud.
¿Qué información genética se guarda de un paciente? Actualmente el componente principal de la capa de datos es la base de datos germinal (Figura 2). Esta base de datos permite almacenar información relacionada con el estudio genético realizado y las variantes genéticas del paciente junto con su significancia clínica. Una variante genética es un cambio en la secuencia de ADN (con respecto al genoma de referencia, o secuencia considerada “normal”) que puede o no afectar al producto de un gen.
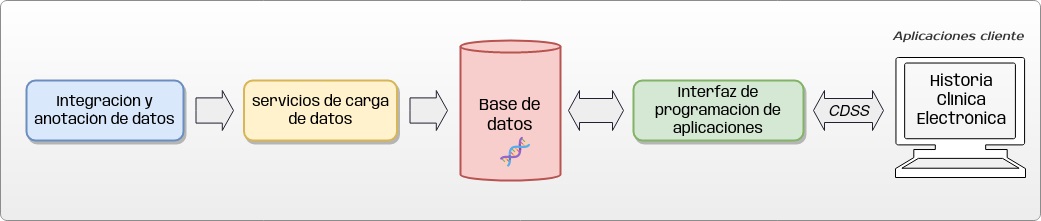
Figura 2: Arquitectura de alto nivel de la solución bioinformática implementada en el Hospital Italiano de Buenos Aires
Debido a la gran cantidad y variedad de estudios genéticos disponibles, no todos los indicados para pacientes se realizan en el laboratorio de secuenciación de la institución. Por esta razón, la solución incorpora una aplicación web de desarrollo propio, que permite la carga de estudios de otros laboratorios junto a las variantes identificadas a la base de datos germinal, utilizando formularios que realizan inferencias y validaciones de datos genéticos en tiempo real, con el fin de simplificar la carga.
Otros componentes del sistema son: 1) el servicio de carga automática de información a la base de datos, que incorpora la información genética del paciente cada vez que se reporta un estudio genético, integrándose directamente con el Sistema de Información del Laboratorio (LIS) y los equipos secuenciadores de ADN, y 2) la API genética, que contiene las funciones bioinformáticas de alto nivel, como por ejemplo, realizar consultas predefinidas a la base de datos y procesar e integrar los resultados con bases de datos genéticas bioinformáticas públicas. Este servicio retorna los resultados utilizando el estándar de interoperabilidad Fast Health Interoperability Resources (FHIR), lo que permite integrarse a cualquier sistema de información en salud. Para su implementación en el HIBA, las funciones son utilizadas por el servidor CDSS para obtener la información genética de los pacientes.
Los beneficios
Con respecto a las reglas genómicas implementadas en el CDSS, las mismas pueden ser clasificadas en dos tipos: reglas de derivación para asesoramiento genético y reglas de seguimiento de pacientes con alteraciones genéticas previamente identificadas. Las reglas de derivación para asesoramiento genético son sugerencias que aparecen en pacientes con riesgo aumentado de padecer alguna variante patogénica heredable. En la versión actual, las reglas incorporadas sugieren al médico una derivación al Programa de Cáncer Hereditario del HIBA, cuando el sistema identifica diagnósticos de cáncer de mama en mujeres menores de 46 años, cáncer de mama en hombres, cáncer de ovario en pacientes mujeres o cáncer de páncreas en pacientes de cualquier género.
Respecto de las reglas de seguimiento, las mismas se refieren a recomendaciones de cuidado para pacientes con riesgo genético aumentado, dirigidas al médico de cabecera. Por ejemplo, en pacientes con variantes patogénicas en los genes BRCA1 y/o BRCA2, que tienen mayor riesgo de padecer melanoma cutáneo, se recomienda una interconsulta anual con dermatología para un examen completo en búsqueda de dichas lesiones, así como asesoramiento acerca de cuidados preventivos. El sistema está diseñado para escalar fácilmente las patologías consideradas y las reglas incorporadas al CDSS.
Cada uno de los componentes que forman este sistema se encuentra implementado localmente en los servidores del HIBA. De todas maneras, los componentes de la solución son lo suficientemente genéricos como para poder ser implementados en cualquier institución de salud debido al uso de estándares de interoperabilidad y nomenclatura genética y de modernas tecnologías de desarrollo, almacenamiento e implementación. El punto de adaptación de mayor esfuerzo se enfocaría en la integración con los sistemas propios de cada institución (LIS y sistemas de interpretación propios de cada laboratorio de secuenciación).
Actualmente, el equipo se encuentra trabajando junto al servicio de Anatomía Patológica del HIBA en la implementación de una segunda base de datos genética, con información de la línea somática de pacientes con algún tipo de cáncer y en la incorporación de una nueva regla para el CDSS con recomendaciones vinculadas a variantes genéticas patogénicas (formas hereditarias de cáncer) en pacientes con un cáncer colónico diagnosticado antes de los 50 años.
En búsqueda del reconocimiento académico internacional, el año pasado (2022) fue publicado un artículo relacionado específicamente con el análisis de las reglas genéticas en el CDSS. Y recientemente, en febrero de 2023, un artículo científico sobre la arquitectura de ésta solución fue aceptado para participar en la Conferencia Internacional de Informática Médica (MedInfo 2023) organizada por la Asociación Internacional de Informática Médica